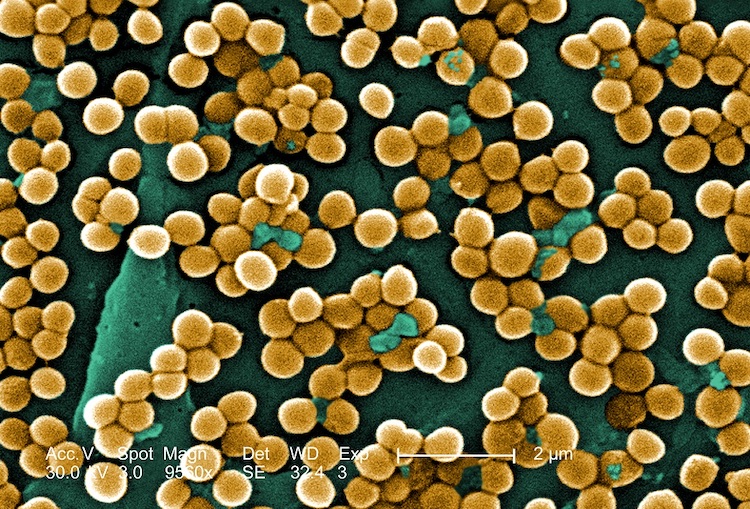
深層学習と呼ばれる人工知能(AI)の一種を用いて、マサチューセッツ工科大学(MIT)の研究者らは、毎年アメリカ合衆国で10,000人以上の死亡を引き起こす耐薬性細菌のクラスを殺すことができる化合物を発見しました。Nature誌の論文で、これらの化合物がメチシリン耐性黄色ブドウ球菌(MRSA)を実験室の培養皿で、そしてMRSA感染のマウスモデル2つで殺すことができることが示されました。これらの化合物は人間の細胞に対して非常に低い毒性を示すため、特に良好な薬剤候補です。12月20日に掲載されたこの論文のタイトルは「Discovery of a Structural Class of Antibiotics with Explainable Deep Learning(説明可能な深層学習による抗生物質の構造クラスの発見)」です。
新しい研究の主な革新点は、研究者らが、深層学習モデルが抗生物質の有効性を予測するために使用している情報の種類を理解できたことです。この知識は、モデルによって同定されたものよりもさらに優れた薬剤を設計するのに役立つ可能性があります。
「ここでの洞察は、モデルが特定の分子が良い抗生物質になるだろうと予測するために何を学んでいるかを見ることができたことでした。私たちの研究は、化学構造の観点から、これまでにない方法で、時間効率良く、リソース効率良く、機構的に洞察に富んだフレームワークを提供します」と、MITの医工学・科学研究所(IMES)および生物工学部門のTermeer医工学・科学教授であるジェームズ・コリンズ博士(James Collins)は述べています。
フェリックス・ウォン博士(Felix Wong)、IMESおよびMITとハーバードのブロード研究所のポスドク、そしてエリカ・ジェン博士(Erica Zheng)、コリンズに指導された元ハーバード医科大学院の大学院生は、この研究の主要著者です。この研究は、7種類の致命的な細菌に対して新しい抗生物質クラスを発見することを目指す、コリンズが率いるMITのAntibiotics-AIプロジェクトの一環です。
説明可能な予測
MRSAは、毎年アメリカ合衆国で80,000人以上の人々を感染させ、しばしば皮膚感染症や肺炎を引き起こします。重症の場合は、致命的な可能性のある血流感染症である敗血症に至ることがあります。
コリンズと彼の同僚たちは、過去数年間、新しい抗生物質を見つけるために深層学習を使い始めました。彼らの仕事は、病院でよく見られる細菌であるアシネトバクター・バウマニに対する潜在的な薬剤、および多くの他の耐薬性細菌に対する潜在的な薬剤をもたらしました。
これらの化合物は、抗菌活性に関連する化学構造を識別することを学習できる深層学習モデルを使用して同定されました。次に、これらのモデルは他の数百万の化合物を精査し、強力な抗菌活性を有する可能性のあるものの予測を生成します。
このような検索は実りあるものでしたが、このアプローチの制限の一つは、モデルが「ブラックボックス」であるということです。つまり、モデルが予測に基づいている特徴が何であるかを知る方法がありません。科学者がモデルがどのように予測を行っているかを知っていれば、追加の抗生物質を特定または設計することがより容易になるかもしれません。
「この研究で私たちが目指したのは、ブラックボックスを開くことでした」とウォンは言います。「これらのモデルは、神経接続を模倣する非常に大きな数の計算から構成されており、その下で何が起こっているのかを本当に知っている人はいません。」
まず、研究者らは、大幅に拡張されたデータセットを使用して深層学習モデルをトレーニングしました。彼らは、MRSAに対する抗生物質活性の約39,000の化合物をテストすることによって、このトレーニングデータを生成し、その後、このデータと化合物の化学構造に関する情報をモデルに入力しました。
「基本的に任意の分子を化学構造として表現できますし、その化学構造が抗菌性を持つかどうかもモデルに伝えます」とウォンは言います。「モデルは、このような多くの例でトレーニングされます。その後、新しい分子、新しい原子と結合の配置を与えると、その化合物が抗菌性であると予測される確率を教えてくれます。」
モデルが予測を行う方法を理解するために、研究者たちはモンテカルロツリーサーチというアルゴリズムを適応させました。このアルゴリズムは、AlphaGoなどの他の深層学習モデルをより説明可能にするのに役立ってきました。この検索アルゴリズムにより、モデルは各分子の抗菌活性の推定値だけでなく、その活性に貢献している可能性のある分子のサブ構造に関する予測も生成することができます。
有効性の候補薬をさらに絞り込むために、研究者らは3つの異なるタイプの人間の細胞に対する化合物の毒性を予測するために、追加の深層学習モデルを訓練しました。この情報と抗菌活性の予測を組み合わせることにより、研究者らは微生物を殺しながら人体に最小限の悪影響を及ぼす化合物を発見しました。
このモデルのコレクションを使用して、研究者らは商業的に利用可能な約1200万の化合物をスクリーニングしました。このコレクションから、モデルはMRSAに対して活性があると予測される5つの異なるクラスの化合物を、分子内の化学サブ構造に基づいて同定しました。
研究者らは約280の化合物を購入し、実験室の皿でMRSAに対してテストし、同じクラスからの2つが非常に有望な抗生物質候補であることを発見しました。MRSAの皮膚感染症およびMRSAの全身感染症の2つのマウスモデルでのテストでは、これらの化合物のそれぞれがMRSAの集団を10分の1に減少させました。
実験は、これらの化合物が細胞膜を維持する細菌の能力を乱すことによって細菌を殺すことを示しました。この勾配は、ATP(細胞がエネルギーを蓄えるために使用する分子)を生成する能力を含む、多くの重要な細胞機能に必要です。コリンズの研究室が2020年に発見した抗生物質候補であるハリシンは、類似の機構によって機能しますが、グラム陰性細菌(細胞壁が薄い細菌)に特有です。MRSAはグラム陽性細菌であり、細胞壁が厚いです。
「この新しい構造クラスがグラム陽性病原体に対して活性があり、細菌のプロトン動機力を選択的に散逸させることで、人間の細胞膜に大きな損傷を与えることなく細菌細胞膜を選択的に攻撃しているという強力な証拠があります。私たちの大幅に拡張された深層学習アプローチにより、この新しい構造クラスの抗生物質を予測し、それが人間の細胞に対して毒性がないことを発見することができました」とウォンは言います。
研究者らは、Antibiotics-AIプロジェクトの一環としてコリンズらが開始した非営利団体であるPhare Bioとその発見を共有しました。この非営利団体は現在、これらの化合物の化学的特性と潜在的な臨床使用について、より詳細な分析を行う予定です。一方、コリンズの研究室は、新しい研究の発見に基づいて追加の薬剤候補を設計する作業、およびモデルを使用して他のタイプの細菌を殺す化合物を探す作業を進めています。
「化学サブ構造に基づいて、既にデ・ノボで化合物を設計するための同様のアプローチを活用しています。もちろん、このアプローチを箱から出してすぐに、異なる病原体に対する新しいクラスの抗生物質を発見するために容易に採用することができます」とウォンは言います。
MIT、ハーバード、ブロード研究所の他、論文の貢献機関にはIntegrated Biosciences, Inc.、生物学的にインスパイアされたエンジニアリングのためのWyss Institute、そしてドレスデンのLeibniz Polymer Research Instituteが含まれます。
この投稿は、MITの科学ライター、アン・トラフトンによるニュースリリースに基づいています。
[News release] [Nature abstract]