インフォコム株式会社より研究支援ツールのご案内
タンデム質量分析装置から得られたMS/MSピークリストから、de novoシーケンシングとデータベースサーチの統合解析を実現
PEAKS 統合プロテオミクス解析ソフトウェア

PEAKS 統合プロテオミクス解析ソフトウェア
- ベンダーニュートラルな包括的なプロテオミクスツールで、各社の質量分析計からLC-MS/MS データの直接読み込みが可能
- de novoシーケンシングによる、アミノ酸配列未知のタンパク質を含めたタンパク質の網羅的解析を実現
- de novoシーケンシング・データベースサーチ・翻訳語修飾解析・Mutation解析までシンプルなインターフェース上での簡単操作で実施可能
- 定量解析・3次元のイオンモビリティデータ専用モジュールあり(オプション)
de novoシーケンシング機能では、データベースに依存せずに、サンプル中に含まれるターゲットタンパク質のアミノ酸配列や、翻訳後修飾の解析が可能で、未知タンパク質やデータベースが整っていない種のタンパク質、生体内ペプチドや合成ペプチドなどの解析に有効です。
さらにタンパク質同定(データベースサーチ)や、定量解析など一連のプロテーム解析が可能です。
DDA/DIAともにサポートしており、最新のプロテオーム研究に有用です。
PEAKS 統合プロテオミクス解析ソフトウェアの詳細について
PEAKSの機能
PEAKSでは、以下のフローに沿って解析を行います。
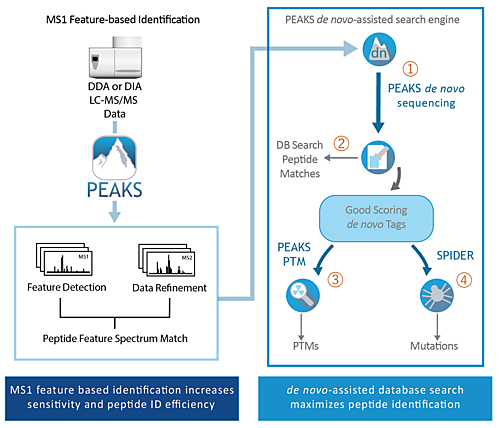
① de novoシーケンシング
de novoシーケンシングは、MS/MSから得られたイオンの質量をもとに、ペプチドのアミノ酸配列(翻訳後修飾含)を算出します。
データベースは全く使用しませんので、データベースに登録されていない配列の解析も可能です。
手動で行うと非常に面倒なde novoシーケンシングも、PEAKSではワンクリックでOKです。

② データベースサーチ
既知タンパク質がターゲットの場合、データベースサーチ機能でタンパク/ペプチドの同定を行います。
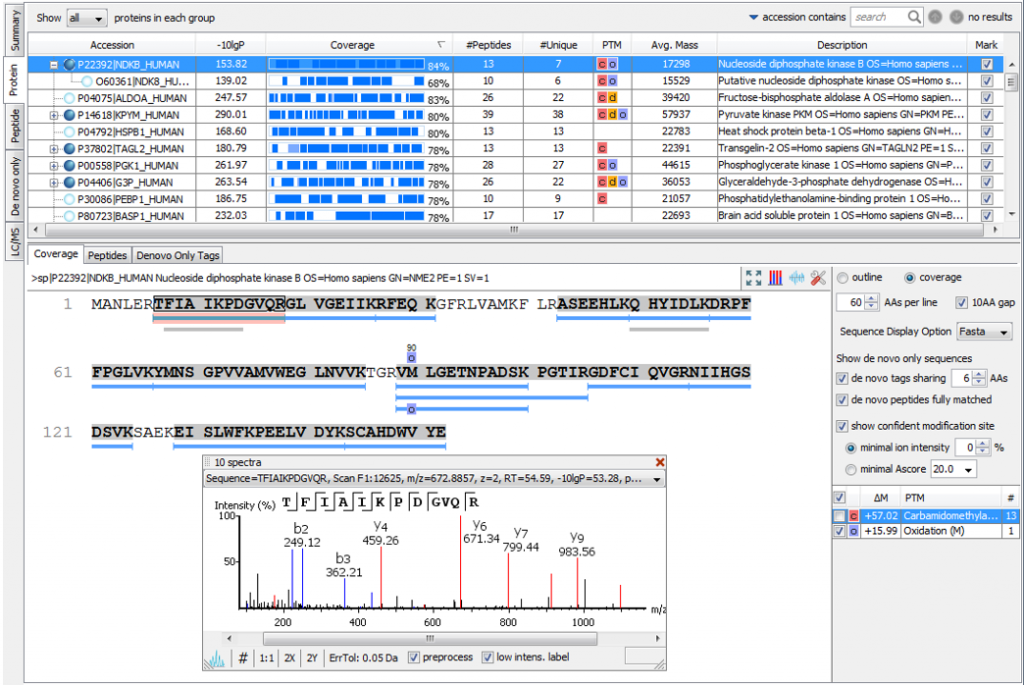
同定結果は、Summary・Protein・Peptide・Feature毎にタブ形式で見やすく設計され、また、グループタンパク(ファミリータンパク)がまとめて表示されるので、タンパク質としての検索スコアが非常に見やすくなっています。
更に未同定のMS/MSに対しては、de novno結果を参照し、一部配列がマッチしているスペクトルなどをチェックすることも可能です。
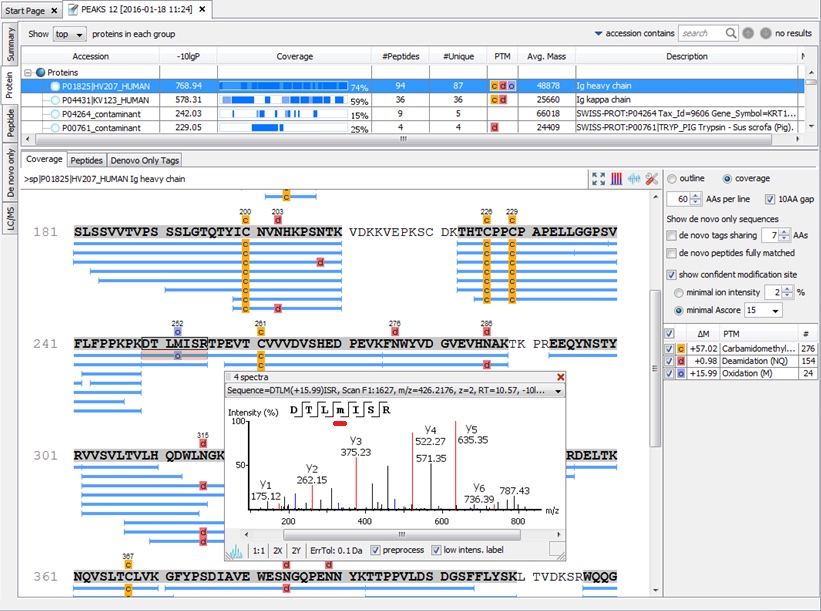
③ 翻訳後修飾 『PTM Finder』
通常、DBサーチの時点では翻訳後修飾を多く設定することができず、そのために未設定のVariable修飾が原因で、未同定になる場合があります。
PEAKSのPTM Finder機能では、未同定のMS/MSかつ同定済タンパク質に対して、網羅的に翻訳後修飾をVariableで再解析を行うことができます。
これにより、全体のカバレージがアップします。
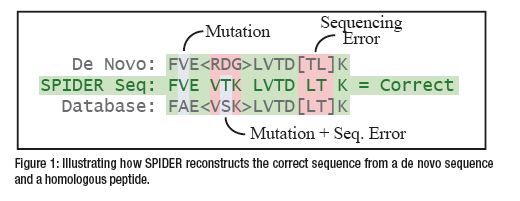
④ Mutation解析 『SPIDER』
データベースサーチや、網羅的な翻訳後修飾サーチでも同定されなかったMS/MSに対しては、ホモロジーサーチ機能を持つSPIDERを適用することで、Mutationを示唆することが可能です。
PEAKS オプション
- 定量解析モジュール:PEAKS Q
PEAKS Qオプションモジュールは、ラベル・ラベルフリー技術の両方を使った実験から、自動的にタンパク質を定量することが可能です。
※Label-free Quantification(LFQ), TMT/iTAQ, SILAC, Custom methods サポート
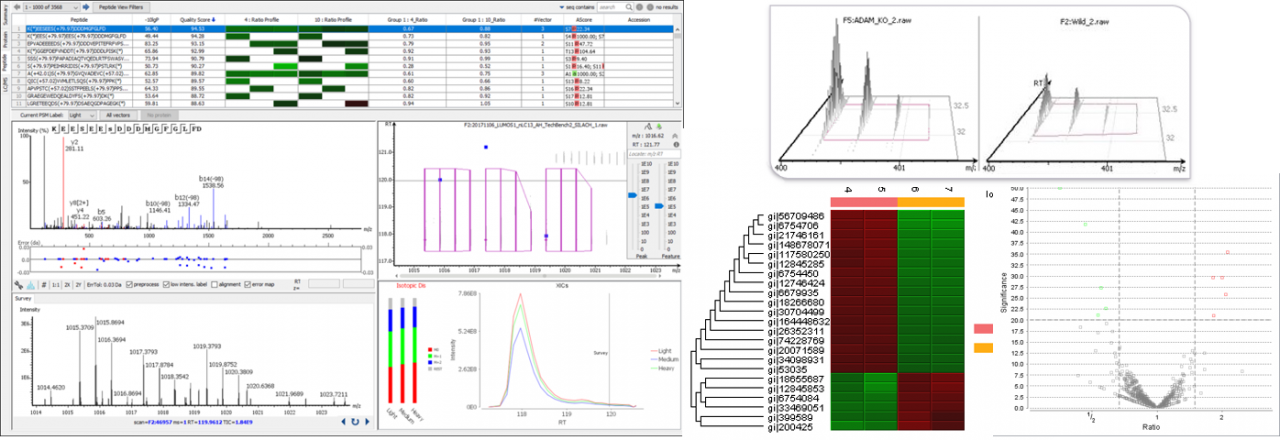
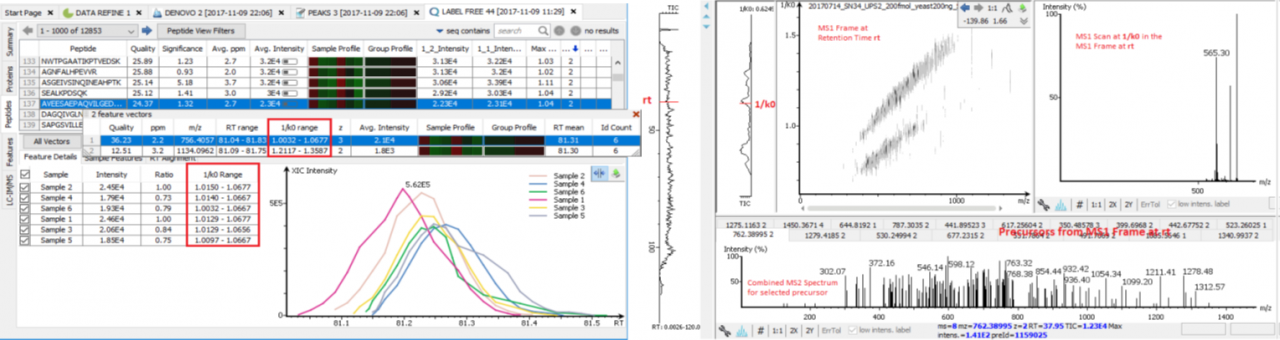
- イオンモビリティデータ専用モジュール 『PEAKS IM』
PEAKS IMオプションモジュールは、Bruker timsTOF Pro/Thermo FAIMS/ Waters HDMSe のイオンモビリティ質量分析装置から得られる3次元データの取込・表示・解析が可能です。
※定量解析(LFQ、TMT/iTRAQ 対応)まで実施する場合はPEAKS Qも必要
読み込み可能なデータフォーマット
- Waters社:Q-TOF(raw)
- Thermo社:Ion Trap / Orbitrap (raw)
- Shimadzu社:AXIMA-CFR(run)
- VarianMS社:FTICR / Ion Trap(sms / xms)
- Agilent社:Ion Trap / Q-TOF(yep / d)
- SCIEX社:TripleTOF / QSTAR / QTRAP /TOF TOF(wiff2 / wiff / t2d )
- Bruker社:Q-TOF / Ion Trap / timsTOF Pro / TOF TOF(baf / yep / tdf / lift)
- 共通フォーマット: .DAT (AB converter) / .DTA (Sequest) / .MGF (Mascot) / .mzData (HUPO format) / .mzML (including mzIdentML) / .mzXML / .PepXML / .PKL (MassLynx) / .XML (mzData schema) / .XML (mzXML schema)
※その他はお問い合わせください。
稼働環境
- OS: Windows 10(64bit) 日本語/英語どちらも可
- CPU: Quad-core processors (Core i7/i9 or Xeon processors with total 16 thrads以上推奨)
- メモリ: 16GB以上(32GB以上推奨)
- ディスクスペース:1TB以上
PEAKS 統合プロテオミクス解析ソフトウェアのお役立ち資料

PEAKS カタログ
ダウンロードURLをリクエスト

PEAKS ご紹介資料
ダウンロードURLをリクエスト

インフォコム取扱製品ラインナップ_2021年版
ダウンロードURLをリクエスト
ご興味がある資料がございましたら、お気軽にリクエスト送信してください。
PEAKS 統合プロテオミクス解析ソフトウェアに関するアンケートのお願い
ご入力いただいた個人情報は、インフォコム株式会社より製品やセミナーのご案内をお送りする際に利用させていただきます。
