インフォコム株式会社より研究支援ツールのご案内
免疫レパトア解析精度の向上に
MIGEC 免疫レパトア解析データ用エラー修正ソフトウェア

- 次世代シーケンス(NGS)データのサンプル毎の分類
- UMI(Unique Molecular Identifier)シーケンスの抽出
- VDJ領域のマッピング、CDR3領域の抽出、ヒトおよびマウス免疫受容体クロノタイプのアセンブリ
- CDR3シーケンスのホットスポットエラーの追加フィルタリング
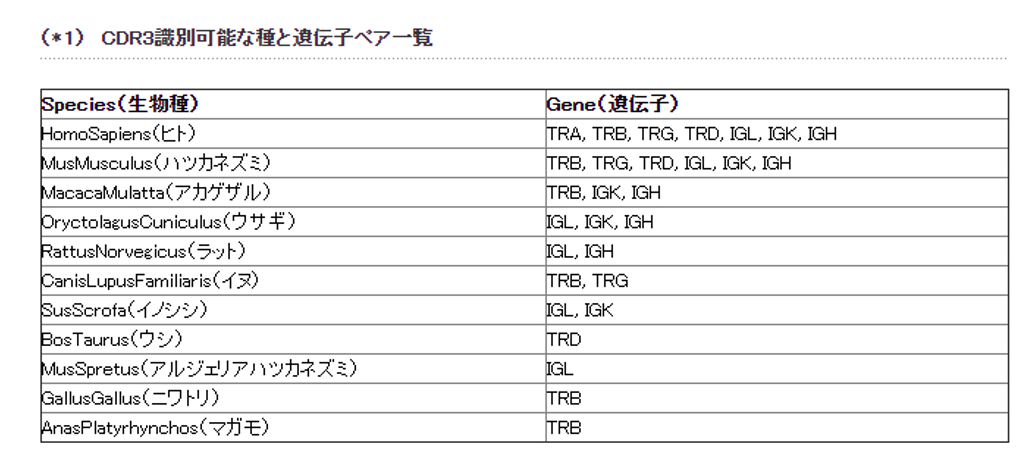
- CDR3の識別を可能にする生殖系列配列(IMGTに基づく)を持つ種と遺伝子のペアをサポート(*1)
- 同一の分子識別子でリードをグループ化することにより、コンセンサス配列のアセンブリが可能
MIGECは、分子バーコード技術(MIG)を使用して生成された免疫レパトアシーケンスデータのエラー修正を容易にし、ロバストでエラーのない、高精度な結果を提供するソフトウェアです。
ハイスループットシーケンスによるTCR/BCRレパートリーの詳細なプロファイリング精度は、PCR増幅エラーとNGSシーケンスエラーの蓄積によって損なわれることが課題となっています。
MIGECは独自のホットスポットエラー修正アルゴリズムによって、複雑な免疫レパートリーが持つ本来の多様性を維持しつつ、ほぼ全てのエラー修正を行うことが可能です。
製品の詳細
CDR3識別可能な種の遺伝子ペア 一覧
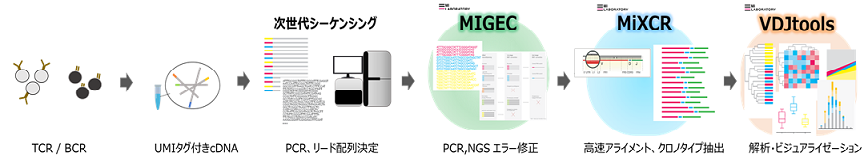
MiLaboratories製品ファミリーのレパトア解析フロー
MiLaboratories社製品は3つの製品で構成されています。「MIGEC」でNGS(次世代シーケンサー)から得られたシーケンスデータを読み込み、エラー修正を行います。つぎに「MiXCR」でクロノタイプの抽出を行い、「VDJtools」で結果のビジュアリゼーションや統計解析を行います。それぞれ単独でも利用可能です。
MIGECのエラー修正アルゴリズム
MIGECは、cDNA合成の段階で導入されたユニークな分子識別子に依存し、下図の独自のホットスポットエラー修正フローに基づいて、シーケンスデータからほぼすべての実験エラーをフィルタリングします。

入力ファイル
MIGECの解析には下記入力ファイルが必要です。
- FASTQ形式ファイル
- UMI配列情報が記載されたファイル
※本ソフトは、Illumina社 MiSeq™ および HiSeq™ を使用して配列決定された、ライブラリー向けに設計されています。
稼働環境
- OS:下記記載の JAVA が動作する Windows、Linux、MAC OS X
- Java Runtime Environment バージョン 8 以上
- グラフの出力には R の実行環境を必要とします(v 3.1.0 でテスト済)
* R は MIGEC 及び VDJtools の動作に必要です。
文献情報
Shugay M et al. Towards error-free profiling of immune repertoires. Nature Methods 11, 653-655 (2014) doi: 10.1038/nmeth.2960.
*姉妹製品「MiXCR」、「VDJtools」と連携可能です。
お役立ち資料

MiLaboratories社3製品(MiXCR/MIGEC/VDJtools)カタログ


